产学研实践活动 | 浙大博士科研前沿分享—TCR测序技术进展综述
发布时间:2025-02-19 11:55 | 点击次数:9971
本期分享由艾沐蒽-浙江大学钱塘智慧城社会实践基地的浙江大学医学院2021级叶茅博士和2022级章港博士供稿
T细胞表达的T细胞受体(TCR)由TCRα链和TCRβ链组成,负责识别与主要组织相容性复合物(MHC)结合的抗原,并驱动针对病原体和癌症的抗原特异性适应性免疫反应。每个人的TCR库非常多样化,这种多样性使得T细胞能够识别广泛的外来抗原,但也为使用传统方法分析带来了挑战。近期的研究开发了高通量测序技术,这些技术不仅能够识别TCR序列,还可以通过实验和计算工具分析其抗原特异性,并将TCR与单个细胞的转录和表观遗传状态表型进行配对。本综述主要介绍了这些技术的进展,并描述了它们在揭示T细胞介导的免疫反应中所发挥的作用。
背景
T细胞是适应性免疫系统中的关键效应细胞,它们在抵御病原体和癌症的同时,也维持着免疫自我耐受。T细胞能够执行多种功能,这种广泛的功能来源于个体内T细胞表型和抗原特异性的多样性。T细胞可以表现出各种不同的表型,从高度细胞毒性的效应T细胞到抗炎的调节性T细胞。这种表型的特化主要由每个T细胞表面特定的T细胞受体(TCR)驱动,TCR负责介导抗原识别和T细胞反应,同时为T细胞群体的多样性提供了另一种表现形式。TCR由两条链组成,分别是TCRα链和TCRβ链,这些链条是通过多个可变基因(V)、多样性基因(D)(仅在β链中存在)、连接基因(J)和恒定基因(C)片段的组合体细胞重排生成的。除了这些重组过程,还存在非模板化的核苷酸添加或删除,使得TCR库具有理论上的多样性,约为2×1019个独特的TCRαβ对。然而,由于选择压力和T细胞数量的限制,个体内的实际多样性可能会低于这一数值。特别是在TCR的高变互补决定区3(CDR3),这种巨大的多样性使得T细胞能够识别大量由MHC(pMHC)复合物呈现的外源和自体肽段。通过分析TCR库,可以更好地理解T细胞反应的几个方面。由于在个体中通过体细胞重组产生完全相同的V(D)J重排的概率非常低,TCR序列可以作为T细胞克隆的独特标识符。这一特性可用于测量:(1)抗原驱动的T细胞克隆扩展,作为抗原特异性和反应的指标;(2)T细胞反应的纵向克隆动态性和异质性。此外,对单个细胞中的TCR和细胞表型进行联合评估,可以提供有关T细胞分化途径的信息,以及TCR序列和特异性在T细胞选择、激活和表型特化中的作用。这些信息不仅有助于理解免疫介导疾病的病因和病理,还能帮助设计治疗策略。通过在不同时间点跟踪患者的TCR多样性,可以揭示与治疗反应或其他临床相关特征相关的T细胞克隆动态模式,将克隆信息与细胞表型相结合,还可以识别与疾病相关的细胞类型。
早期的TCR分析方法虽然揭示了TCR库的一些基本特征,但通常只能捕捉部分序列信息。例如,CDR3光谱分析通过电泳分离PCR扩增子来测量CDR3序列长度分布,用于初步估算人类和小鼠的TCR多样性。然而,这种方法仅针对特定的V-J重排,无法直接读取序列信息,而序列信息对于确定T细胞的克隆性和特异性至关重要。另一种方法依赖于流式细胞术和抗-TRBV单克隆抗体,基于TCRβ V基因的使用情况提供库多样性的见解,但同样未能捕捉到序列信息或CDR3的连接多样性。随着基因组测序技术的发展,早期的TCR测序方法通过分子克隆和Sanger测序在核苷酸水平捕获TCR,从而能够更精确地测量CDR3的多样性。这些方法广泛应用于免疫介导疾病、感染和癌症的TCR库研究,揭示了某些TCRβ基因的疾病特异性扩展。这些方法还首次估算了TCRα和TCRβ序列的多样性,并证实初始T细胞库的高度多样性,而记忆T细胞群体仅占个体总TCR库多样性的一小部分。然而,这些技术的通量较低(每次实验只能测量10到1,000个TCR),因此难以对样本或群体中的TCR多样性进行全面采样。
在过去的二十年里,随着新一代测序技术的普及,TCR检测的敏感性和应用TCR发现工具的便捷性大大提高,每次实验可捕获10^3到10^6个TCR,并在其细胞环境中分析TCR序列。本综述讨论了在大规模和单细胞分辨率下研究TCR库的高通量方法的新进展,以及将TCR序列与抗原特异性、细胞状态和表型配对的技术。特别是这些方法如何深化我们对T细胞克隆动态、分化轨迹及其在健康与疾病中的功能的理解。
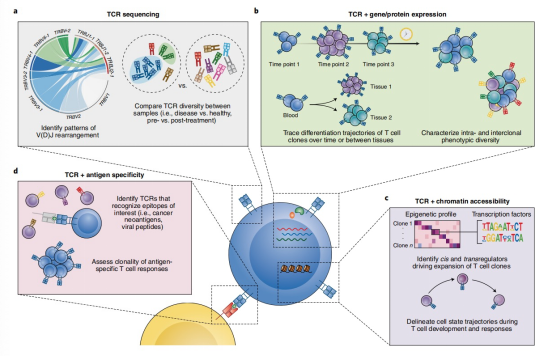
图1. 使用多组TCR测序方法表征T细胞动态
一、TCR测序的方法
使用高通量测序技术对TCR库进行分析使得在单次实验中能够分析数百万个T细胞。这些方法通常采用以下两种扩增策略之一:(1)多重PCR或 (2)5′端互补末端快速扩增(5′ RACE)。
1.1 多重PCR
由于TCR V基因具有高度的序列多样性,单一对引物不足以捕捉所有TCR转录本。为了解决这一挑战,最早开发的针对TCR的测序方法之一是多重PCR反应。多重PCR方法使用一组针对所有已知V基因的前向引物和一组针对J或C区域的反向引物,具体取决于起始材料是基因组DNA(gDNA)还是互补DNA(cDNA)。在这种引物扩增策略中,从T细胞中分离gDNA或RNA,利用这些引物集进行多轮PCR扩增,这些引物还包含通用引物序列,以便后续在高通量平台上进行测序。多重PCR方法的使用带来了对人类TCR多样性的许多早期见解。例如,Robins等人对CDR3β进行了深度测序,展示了在稳态下TCR库对某些V-J重排表现出强烈的偏倚。他们还观察到个体之间的naive CDR3β重叠显著大于随机期望。这些结果表明,TCR库的生成并非随机,而可能偏向特定的重排,可能是在胸腺发育过程中通过T细胞选择或共同的抗原经历被修剪过的。由于这种方法在测序前依赖于多轮PCR,可能引入测序偏倚和错误,从而扭曲TCR多样性的测量或生成人工TCR序列。然而,存在几种策略可以缓解这种偏倚。例如,由于cDNA转录本已经剪接,可以使用较少的反向引物来靶向C区域,从而减少来自多重J引物的PCR扩增偏倚。相反,从gDNA开始则省去了逆转录步骤,这也最小化了在cDNA合成过程中引入的错误。此外,使用合成TCR分子靶向多重引物,可以在多重PCR前后量化模板,从而优化引物浓度并校正扩增偏倚。
1.2 5′ RACE
另一种用于TCR测序的方法是基于5′ RACE。该方法首先使用具有末端转移酶活性的逆转录酶将RNA逆转录为cDNA,末端转移酶在cDNA的3′末端添加无模板的C核苷酸。随后,一个包含互补聚(G)序列的模板切换寡核苷酸(TSO)与这些无模板区域结合,使逆转录酶能够切换模板并继续扩展cDNA,直到TSO的末端,该末端包含一个通用适配子序列。因此,仅需一对靶向5′适配子和恒定区域的引物即可扩增所有TCR重排。尽管5′ RACE策略需要的PCR轮次较少,从而减少了多重PCR方法中常见的扩增偏倚,但这些方法仍然可能受到PCR、模板切换或测序过程中的错误影响。
1.3 错误校正策略用于TCR扩增方法
虽然这些TCR测序策略旨在提供TCR库的准确表示,但如上所述,它们仍受到扩增偏倚和测序错误的影响。引入分子标识符(UMIs)提供了一种纠正这些错误的策略。UMIs是在cDNA合成过程中附加的随机DNA序列,用于标记每个cDNA分子。这种策略有两个主要优点:(1)通过对每个UMI进行一次计数,可以恢复样本中TCR序列的原始分布,从而更准确地量化TCR克隆频率;(2)通过将标记为相同UMI的读取结果分组,可以通过共识推断真实的TCR序列,从而校正PCR和测序错误(尽管在反转录过程中引入的错误仍未解决)。由于TCR可能仅在几个核苷酸上有所不同,这一校正步骤可以区分真实的TCR变异和错误引起的伪造多样性,从而提供对TCR库多样性的更准确估计。然而,这种准确性可能会以较低的灵敏度为代价,因为低频克隆可能由于每个UMI的读取覆盖不足而被过滤掉。为生成更精确的克隆型频率测量,已经开发了如基于每个核苷酸的质量分数的错误校正算法。
1.4 TCR靶向方法的比较
鉴于TCR测序策略之间的技术差异,用户在分析TCR库时应考虑几个因素。首先,起始材料的可用性可能决定了适用的方法。所有方法均可使用RNA,因为与基因组DNA(gDNA)相比,RNA模板的拷贝数更高,使得TCR序列的检测更加灵敏。然而,如果RNA质量较差,例如来自化学保存的档案组织样本,则应选择gDNA多重PCR方法,因为5′ RACE方法需要高质量的RNA作为起始材料。其次,TCR分析的目的也可能影响方法的选择。例如,RNA测量不能直接与细胞数量相关联。因此,如果目标是定量T细胞群体的克隆扩增,gDNA方法更为适宜,因为每个细胞中有一个基因组拷贝的TCR。类似地,由于多重PCR使用的引物集针对V基因的不同区域,因此不一定能捕获跨越整个V(D)J区域的全长TCR序列。这对于分析包含最高序列变异性的CDR3可能已经足够。然而,如果回答特定生物学问题需要评估其他区域的变异性,如CDR1和CDR2,则5′ RACE方法更为合适。此外,这些TCR测序方法中每个步骤的技术特征对最终的TCR库数据及其解释具有重要影响。在最近的一项关于多重PCR和5′ RACE TCR测序方法的比较研究中,Barennes等对同一批T细胞样本上的九种TCR测序方案进行了基准测试,以评估它们的可重复性、可复制性和灵敏度。他们检测到了方法特有的TCR库特征,这些特征在重复实验中基本一致,表明每种方案在捕获TCR库时都存在独特的偏倚。通过将TRB使用情况与流式细胞术频率进行比较,他们观察到,与gDNA基于的多重PCR或5′ RACE方法相比,基于RNA的多重PCR方法对特定V基因的偏倚更强,这可能反映了由于RNA转录本丰度差异导致的扩增偏倚。无论方法如何,获取代表真实生物学TCR库的测序结果也高度依赖于起始材料的数量,特别是对于检测稀有TCR序列而言。如果研究的目标是捕获高程度的TCR多样性或在高度多样化的样本中检测稀有克隆,那么使用非UMI的5′ RACE方法可能是最佳选择,因为它们在捕获稀有克隆时比基于UMI的方法更为灵敏,尤其对于TCRα链。相反,如果目标是构建代表性的TCR克隆结构或根据扩增水平识别感兴趣的TCR,那么5′ RACE结合UMI校正可能最为合适,因为与其他方法相比,它能更忠实地再现TCR克隆型频率,尽管可能需要多个重复实验或更高的测序深度来一致性地捕获低频克隆。
二、高通量TCRαβ配对捕获方法
捕获配对的TCRαβ序列能够更准确地解析TCR库中的克隆结构,并且对于评估TCR功能和抗原特异性至关重要。这通常通过以下两种方法实现:(1)使用基于组合数学的策略进行TCRαβ配对的计算推断,(2)对单个细胞同时进行TCRα和TCRβ测序。
2.1 TCRαβ配对的组合推断
一种检索配对TCRαβ库的方法依赖于组合数学。在这种被称为pairSEQ的检测方法中,T细胞被随机分布到一个平板上,使得每个孔中包含一组标记有相同孔特定DNA条形码的细胞。在将样本汇总进行测序后,具有相同条形码集合的TCRα和TCRβ序列会被计算匹配,并推断为一对,因为由于TCR重排的组合性质,两种克隆共享相同孔的可能性非常低。在Lee等人开发的类似方法中,CDR3αβ对是通过对按孔分组的批量测序克隆使用重复采样策略进行计算推断的。这些基于频率的配对方法的一个局限是只能解析扩增的克隆,因此这些检测方法不适合发现稀有的TCRαβ序列。
2.2 单细胞TCR测序技术
近年来,单细胞分离技术的进步使得利用最初为批量群体开发的TCR扩增策略捕获配对TCRαβ链信息成为可能。最早用于单细胞配对TCRαβ分析的方法依赖于激光微切割和微量移液等显微操作技术来分离单个T细胞,然后进行多重PCR和Sanger测序。虽然这些方法在最初的配对TCRαβ库特征描述中发挥了重要作用,但它们耗时且在效率和通量上都有限。目前,单细胞分离的常用方法是荧光激活细胞分选(FACS)。这种方法的优势在于可以根据表面标记的表达富集感兴趣的细胞群体,而不是对样本中的所有T细胞进行测序,这在分析稀有亚群时特别有用。此外,该方法提供的高读取质量和覆盖度(约每条链1000条读取)能获得高置信度的TCR序列,这对于分析克隆多样性至关重要。在此方法中,细胞被用荧光抗体标记表面蛋白,并个别分选到微孔板中,从而一次性分离出数百个单细胞。然后,使用如多重PCR等TCR靶向策略对TCR链进行逆转录和扩增。使用这一策略的初步研究揭示了人类和小鼠在病毒感染中抗原特异性CD8+ T细胞反应的克隆多样性的首次描述,并发现了远高于以前所知的双等位TCRα表达频率。最近对这种FACS基础分离方法的改进包括通过PCR条形码策略结合高通量测序。例如,在Han等人描述的方法中,在嵌套TCR扩增后,通过PCR扩展步骤引入了每个孔特有的寡核苷酸条形码。这些条形码保留了转录本的细胞来源,从而使样本能够在下一代测序仪上进行汇总测序,同时保持TCRαβ配对。另一种对单个细胞进行配对TCRαβ测序的策略是通过细胞基于乳液的PCR方法。这些方法中,单个细胞被捕获在含有TCR引物和逆转录PCR(RT-PCR)试剂的水油乳液中。经过封装和细胞裂解后,T细胞会进行重叠扩展RT-PCR(OE-PCR),使用针对C区的引物以及一组包含互补序列的V区引物,这些互补序列能够在乳液中连接TCRαβ转录本。这些融合产物包含了两个链,可以在保持原生TCRαβ配对的情况下进行测序。Munson等人开发了一种乳液OE-PCR方法,以识别在乳腺癌患者中共享但在健康样本中缺失的肿瘤特异性TCRαβ克隆,提供了抗肿瘤T细胞反应可能也由共享抗原驱动而不仅仅由患者特异性新抗原驱动的证据。微流控平台通过在微孔或微滴中分隔单个细胞,显著提高了单细胞分离的通量,这使得它们在过去几年开发的许多单细胞TCR测序方法中得到了广泛应用。在基于孔的方法中,细胞以低密度加载到纳升级别的孔阵列中,通过重力将单个细胞限制在纳米孔内。另一种方法是基于微滴的方式,细胞在载体油填充的芯片微通道中以一定的流速和压力流动,从而使大多数微滴中含有单个细胞。最近的一次OE-PCR方法使用了一种高通量微滴微流控设备,在单次实验中捕获了数百万个T细胞,从而使作者能够对15名健康个体(包括6对同卵双胞胎)的TCR库进行分析。利用迄今为止收集的最大配对TCRαβ序列数据集,他们展示了无关个体之间的TCR库重叠程度远低于以往单链研究的估计,这突显了配对TCRαβ数据在提供有意义的库多样性见解中的价值。
此外,作者发现V基因和J基因的使用在双胞胎之间的相关性显著高于无关个体,表明遗传因素在胸腺选择中发挥了作用,这可能是由于在相同MHC分子上呈递抗原的影响。
三、单细胞方法用于配对TCR与T细胞状态和功能
尽管以单细胞分辨率捕获TCR序列信息是一项重要的技术进展,但在细胞状态信息的背景下研究TCR库可以提供更全面的T细胞在免疫反应中功能和生理作用的视角。为此,多个研究小组开发了将TCR测序与其他技术相结合的方法。一类方法能够同时测量单个细胞中的TCR和转录组特征。这可以通过以下两种方式实现:(1)从单细胞RNA测序(scRNA-seq)读取中计算重建TCR链,(2)在结合基因表达特征的情况下,特异性扩增TCR位点。
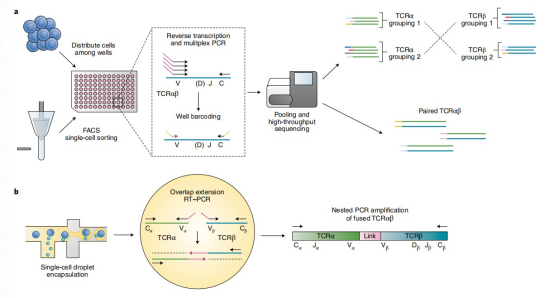
图2.单细胞TCR测序的方法
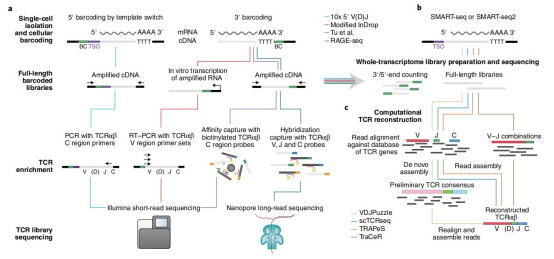
图3. 配对scRNA-seq和TCR测序方法示意图
3.1 通过计算TCR重建将TCR与T细胞表型配对
从scRNA-seq数据中提取TCR信息的计算方法通常依赖于对齐和从头组装步骤的组合,以识别TCR来源的读取并重建TCR链。使用先前scRNA-seq方法进行TCR重建的挑战在于,它们依赖于通过使用poly(A)尾巴作为锚点来扩增3′转录本末端,这样无法捕获位于5′转录本末端的TCR V(D)J区域。为了克服这一问题,新的技术采用全长cDNA扩增和测序策略,其中单细胞通过类似于5′ RACE的模板转换机制进行逆转录。经过片段化和测序后,可以在其表型特征的背景下计算重建并分析个体TCR序列。一种这样的TCR重建算法被称为scTCRseq,它通过使用核苷酸BLAST将读取对齐到V和C基因数据库,从而构建全长配对TCR序列,并将这些读取组装成共识序列。V和C基因的共识序列随后用'N'核苷酸填充,并连接起来,形成一个带间隙的V–C支架,然后对所有读取进行重新对齐,并组装成最终的TCR序列。Stubbington等人开发了一个广泛使用的TCR重建工具TraCeR,该工具使用了类似的过程,但在初始读取对齐时使用了所有可能的V–J组合数据库。另一种TCR重建方法VDJPuzzle则包括额外的对齐和组装步骤,其中第一轮的TCR重建结果被用作更新的数据库,以捕获与参考基因组有显著差异的TCR。与这些需要较长读取(>50 bp)的方法相比,配对末端单细胞TCR重建算法(TRAPeS)通过使用配对读取来识别CDR3来源的读取,并通过迭代动态编程过程扩展CDR3序列,从而实现了对短读取(25–30 bp)的TCR组装。通过将TCR序列与基因表达数据结合,这些计算方法为T细胞克隆表型动态提供了一些初步视角。例如,Stubbington等人应用TraCeR分析了在小鼠沙门氏菌感染期间由SMART-seq在Fluidigm C1系统上生成的CD4+ T细胞scRNA-seq数据。通过将重建的TCR与其细胞转录组配对,作者追踪了T细胞克隆在感染过程中从激活状态到T辅助1(TH1)和效应记忆T细胞状态的分化轨迹,并展示了单一克隆可以采纳多种细胞命运。这一发现证实了之前在病毒感染背景下进行的采纳转移研究中观察到的抗原反应性T细胞命运的异质性,支持了T细胞反应过程中渐进分化的模型。类似地,Ranasinghe等人识别并表征了人类免疫缺陷病毒(HIV)控制者中的非常规MHC II类限制CD8+ T细胞反应,这些HIV控制者能够在没有抗逆转录病毒治疗的情况下维持不可检测的病毒负荷。通过使用TRAPeS,他们证明这些抗原特异性CD8+ T细胞表现出细胞毒性抗病毒表型,并且在每个个体中由单一扩增的TCRβ克隆型主导,这表明非常规II类限制的CD8+ T细胞反应可能在人体病毒感染中扮演了之前未被充分认识的角色,这一发现可能为未来疫苗设计提供新的方向。虽然这些方法利用了之前生成的scRNA-seq数据来分析TCR,但成功的TCR重建依赖于测序深度和TCR位点的表达水平,而这些在细胞间存在差异。因此,由于测序深度不足或起始材料有限而导致的TCR库捕获不完全,可能会影响对克隆性和多样性的生物学解释。
3. 2 通过同时进行RNA和靶向TCR测序以关联TCR与T细胞表型的研究
为生成覆盖度更高的TCR测序库,多个研究团队开发了将RNA分析与单细胞中TCR两条链的靶向扩增相结合的技术。其中一种最早实现这一目标的方法是使用多重PCR策略,同时靶向TCRα和TCRβ链,以及包括细胞因子和对T细胞功能与亚群区分重要的转录因子在内的17个表型基因。这项技术被用于表征结直肠癌中的CD4+肿瘤浸润淋巴细胞(TILs),揭示了FOXP3+RORC+和FOXP3–RORC+ T辅助17(TH17)亚群之间的克隆共享,表明产生白介素(IL)-17的细胞可能源自一个共同的祖先,这些祖先在抗原激活后扩展并分化为不同的表型。这个发现再次支持了CD4+ T细胞在活跃免疫反应中表现出谱系可塑性的概念。此外,这项研究还揭示了FOXP3+ T细胞在肿瘤免疫中的模糊角色,特别是在与IL-17表达相关联的情况下,这种表达已与较差的预后相关联。
后续研究通过将靶向TCR测序与基于微滴的scRNA-seq方法相结合,对全转录组进行分析,进一步提高了配对分析的通量。例如,InDrop方法用于3' scRNA-seq,通过在片段化之前使用特异性针对TRAV和TRBV基因的引物,对扩增的RNA文库的一部分进行修饰,以靶向TCR转录本。Zemmour等人将这种技术应用于从小鼠肠道中分离出的调节性T细胞,发现表达相同TCR的细胞在转录上具有相似性,表明TCR信号传导可以影响T细胞的表型,这一观察后来在其他研究中得到了重复验证。Tu等人开发的类似方法通过使用与C区匹配的生物素化寡核苷酸探针,在cDNA文库中富集TCR转录本。值得注意的是,这种方法与标准的3'条形码平台兼容,包括Seq-Well和Drop-seq,因此无需InDrop所需的特定试剂盒。另一种靶向TCR捕获的方法,被称为通过测序的谱系和基因表达(RAGE-seq),使用特异性针对V和J基因的杂交捕获文库,随后进行长读长Oxford nanopore测序,以捕获全长TCR转录本,而剩余的cDNA文库则通过标准的短读长Illumina测序进行基因表达分析。尽管该方法具有捕获全长TCR序列的优势,但由于纳米孔测序的错误率较高以及需要多种测序技术,TCR回收率相对较低,这可能限制了该方法的广泛应用。最后,5' RNA扩增也被用于同时进行scRNA-seq和TCR测序。在这种方法中,细胞被包裹在含有条形码珠子的微滴中,与3' scRNA-seq方法类似;然而,该方法中的条形码位于与poly(dT)序列相邻的位置,而是在cDNA模板扩展后附加到5'末端的TSO旁边。然后,将全长cDNA汇集并使用靶向PCR引物进行扩增,这些引物与在反转录过程中加入的通用序列匹配。cDNA文库的一部分用于通过PCR和靶向TCR恒定区的引物进行TCR转录本的富集并构建文库,而剩余的材料则直接用于构建基因表达文库。10x Genomics已将这种5'捕获方法商业化,并提供了若干关于T细胞反应的关键见解,特别是在癌症中。在这种方法的首次应用之一中,Azizi等人分析了人类乳腺癌肿瘤中的TIL组分,研究了TCR谱系多样性与T细胞表型多样性之间的关系。通过整合细胞状态和TCR克隆型数据,研究揭示了T细胞活化的连续轨迹,这可以部分用TCR多样性来解释。此外,研究人员观察到T细胞克隆主要局限于表型相关的簇,这些簇表现出类似的环境特征,如无能状态和糖异生,表明细胞状态是由TCR刺激和微环境信号共同驱动的。Yost等人还使用这种方法追踪了基底细胞癌患者在抗PD-1治疗前后的T细胞克隆动态。这项研究发现,治疗后扩展的CD8+ T细胞群体主要由此前在肿瘤中缺失的衰竭克隆组成,表明检查点阻断通过招募新的T细胞克隆而非如先前认为的那样通过重新激活现有的衰竭克隆来发挥作用。
3.3 将TCR与表面蛋白配对分析
对单个细胞的TCR和转录谱同时进行分析,可以为高分辨率的T细胞状态提供有意义的见解。然而,通过蛋白质表达测量进行T细胞表型分析可以补充这一分析,并更清晰地定义T细胞亚群,因为某些细胞类型标志物在转录水平上难以检测到。此外,单细胞蛋白质测量可能更有效地解析异构体的使用,特别是识别表达PTPRC基因异构体的效应和记忆T细胞群体,这些异构体CD45RO和CD45RA在不同水平上表达,以及细胞内蛋白质的活性和修饰,例如关键T细胞转录因子的活性。为此,Mimitou等人最近描述了一种名为扩展CRISPR兼容的细胞转录组和表位索引测序方法(ECCITE-seq),该方法扩展了5′ scRNA-seq和TCR测序方法,以并行捕获细胞表面蛋白、基因表达和TCR谱。这是通过使用与TSO互补的寡核苷酸标记结合DNA条形码抗体来检测蛋白质实现的。在反转录过程中,细胞条形码不仅与抗体衍生的标签,还与微滴内的cDNA模板相关联。扩增和测序后,可以读取这些抗体衍生的标签,并用于在每个细胞中将蛋白质表达与TCR克隆型和RNA表达配对。值得注意的是,这种方法还可以通过使用寡核苷酸结合的pMHC多聚体来评估TCR抗原特异性,我们将在下文中讨论。ECCITE-seq还可以扩展到直接捕获单向导RNA(sgRNA),从而在CRISPR筛选中与RNA、蛋白质和TCR谱并行分析。通过使用额外的反转录引物与sgRNA的恒定3′区域退火,可以捕获sgRNA序列并通过与用于RNA部分类似的模板转换机制将其链接到细胞条形码。概念验证实验表明,ECCITE-seq能够捕获由sgRNA引导的基因干扰引起的mRNA和蛋白质水平的变化。这一方法为未来研究CRISPR介导的干扰如何调控T细胞克隆性和表型提供了一个有前景的途径。
3.4 将TCR与染色质可及性配对分析
一种将TCR序列与其细胞背景进行分析的正交方法是将单细胞TCR捕获与转座酶可及染色质测序(ATAC-seq)相结合,这种技术用于测量全基因组的染色质可及性。这种方法被称为转录索引的ATAC-seq(T-ATAC-seq),涉及单细胞的微流体分离,随后使用Tn5转座酶对基因组DNA进行tagmentation,该转座酶优先插入开放的染色质区域。随后,TCR转录本使用针对C区的引物进行反转录,并通过靶向恒定区和可变区的内引物进行多重PCR扩增,同时ATAC-seq片段进行5′延伸和PCR扩增。该方法使研究者能够探究表观遗传因素(包括顺式作用的DNA元件和反式作用的转录因子)如何驱动T细胞克隆的特异性和扩增。例如,该方法曾用于识别淋巴瘤中恶性克隆性T细胞的表观基因组特征,这一特征此前未被标准的FACS方法检测到。这一发现可能有助于改善当前区分癌性与良性T细胞增殖的诊断能力,并为针对表观基因组的临床治疗提供参考。
四、将TCR序列与抗原特异性映射的策略
将抗原特异性与T细胞克隆性和表型测量相结合,对于理解T细胞反应的驱动因素至关重要。T细胞抗原识别的几个方面使得将TCR序列映射到特异性上成为一个复杂的问题。首先,抗原特异性T细胞的稀有群体频率可能低至百万分之一,使得这些细胞的检测非常困难。其次,由于MHC的多态性、TCR和pMHC的多特异性,以及单个抗原编码的潜在表位种类繁多,导致在解析TCR抗原特异性时面临高度的变异性和复杂性。最后,大多数TCR-pMHC相互作用的亲和力和亲和性较弱,使得选择性分离抗原特异性T细胞群体成为一项艰巨的任务。尽管有多种方法可以评估T细胞的抗原特异性,但我们在此重点讨论能够同时进行TCR测序的策略,并建议对T细胞表位发现感兴趣的读者参考最近的一篇综述。研究抗原特异性T细胞的经典方法之一是使用荧光标记的pMHC多聚体,通过流式细胞术分离能够识别特定肽段的T细胞。然后,可以对这些抗原特异性T细胞群体进行前文讨论的任何TCR测序或单细胞TCR与表型分析方法。尽管这种方法的通量有限,但一些技术进展已显著增加了可同时评估的特异性数量。DNA和磁性纳米颗粒条形码技术的新进展提高了多聚TCR特异性检测的通量,使得可以同时检测超过1,000种肽段特异性。虽然许多这些方法仅用于识别T细胞的抗原特异性,但最近的几个发展使得捕获TCR成为可能,从而能够在抗原特异性T细胞群体中表征克隆性和TCR序列的变异性。其中一种方法是四聚体相关的TCR测序(TetTCR-seq),该方法通过使用荧光标记的DNA条形码pMHC四聚体对T细胞进行染色,并通过FACS将其分选到单细胞孔中。在每个孔内同时进行TCRαβ转录本和DNA条形码的RT-PCR扩增后,细胞条形码可用于在单细胞水平上将TCR序列与其对应的抗原相连接。张等人使用此方法同时筛查了150多对癌症新抗原和野生型肽段,展示了TetTCR-seq在高通量识别功能性新抗原特异性TCR方面的实用性。该方法还可应用于感染、自身免疫和其他免疫介导疾病背景下系统探究T细胞特异性,并可能为过继性T细胞治疗策略提供重要信息。通过微流控技术进一步提高通量,能够同时捕获抗原特异性和TCR序列。一种称为微流控抗原-TCR结合测序(MATE-seq)的方法使用带有磁性纳米颗粒条形码的pMHC四聚体,并将其与光可切割的TCR特异性引物连接,以在微滴中捕获TCR序列和抗原身份。具体而言,T细胞与带有纳米颗粒条形码的pMHC库一起孵育,并通过磁分离纯化。单个细胞被封装在微滴中并裂解后,带有条形码的pMHC暴露在紫外线下,释放出针对TCRαβ C区的RT-PCR引物。由于这些引物与对应于pMHC的DNA条形码相连,即使在汇总和测序后,也可以在单细胞水平上将TCR序列与抗原特异性结合。虽然由于文库构建的限制,目前此方法仅限于少数pMHC四聚体,但未来的改进可能会使分析具有更大抗原特异性阵列的T细胞群体成为可能。
尽管这些进展大大提高了将TCR序列映射到特异性的通量和分辨率,但这些方法仅适用于当目标是表征具有已知抗原特异性的T细胞时,并且感兴趣的肽可以事先指定的情况,在某些情况下这可能并不可行。同样,识别与特定抗原结合的TCR序列的相反问题——识别特定TCR识别的抗原——也是一个挑战。将抗原特异性与TCR序列联系起来的一种仍相对初步的方法是计算表位预测。该方法通过在一组已知的TCR-抗原对上训练监督机器学习模型,并使用该分类器预测新TCR序列的抗原特异性。虽然一些模型能够区分少量表位的TCR特异性,但这些算法目前还无法对完整的TCR库实现高预测准确性,因为模型必须分别为每个表位或表位集合进行训练。改进这些方法将需要比目前通过内部和公共数据库,如McPAS-TCR、VDJdb和免疫表位数据库(IEDB)可获得的更大规模的实验验证的TCR-pMHC对训练集。这可以通过继续收集TCR-pMHC数据来实现,结合上述同时捕获TCR序列和肽特异性的策略,以及使用TCR定向表位发现方法的研究。此外,更好地模拟TCR-pMHC相互作用的三维复杂性和灵活性的算法进展将有助于改进计算表位预测方法。然而,即使有更复杂的算法来改进TCR表位的预测,鉴定与特定抗原特异性反应生理相关的T细胞克隆的问题因TCR的多特异性而变得复杂。由于一个TCR能够识别多个不相关的肽段,这种交叉反应性使得将特异性与体内生物功能精确关联变得困难。鉴于这些挑战,尽管这些机器学习算法可能有助于提名感兴趣的TCR以供进一步研究,但它们可能需要与额外的检测结合,才能为TCR特异性的确定提供完整的解决方案。
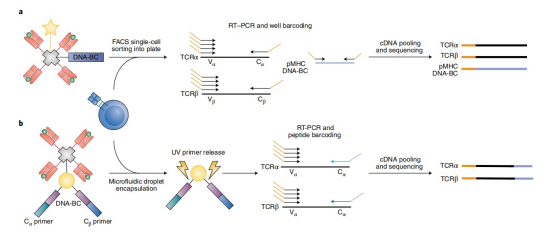
图4. TCR序列与抗原特异性连接的单细胞方法总览
五、TCR测序数据收集的标准
值得注意的是,随着本综述中描述的高通量TCR发现方法的广泛采用,已积累了大量的TCR测序数据。因此,推动TCR测序数据标准化的工作变得尤为重要,以促进数据的可重复性和共享。为此,适应性免疫受体库(AIRR)社区定义了“适应性免疫受体库的最少信息”(MiAIRR)标准,这是一套用于报告抗体和TCR测序研究元数据的指南,涵盖从研究设计和样本处理到数据处理、注释和提交至NCBI存储库的各个方面。此外,建立了一个用于表示注释受体重排的标准化架构和文件格式,这使得对支持该格式的存储库中的数据访问更加顺畅,这些存储库共同组织成AIRR数据共享库。持续努力定义数据标准并鼓励遵守这些指南,对于在研究界内促进一个不断改善的合作生态系统至关重要,这个生态系统通过使用TCR测序技术推动进步。
未来展望
尽管这些TCR测序的进展极大地推动了T细胞领域的发展,但在效率、灵敏度和成本方面,特别是在单细胞方法中,仍然存在改进的需求。目前,基于液滴的测序方法的单个细胞捕获率约为65%。这一低效率限制了研究的可解释性,因为在实验中捕获的细胞可能无法代表起始群体,尤其是在分析外周血液和其他免疫相关器官等大样本时。当前基于液滴的单细胞技术的另一个局限是其最低输入要求约为1,000个细胞,这使得对稀有亚群(如抗原特异性T细胞群体)的分析变得困难。此外,虽然微流控单细胞方法高度并行,并在纳升反应体积中进行,与传统的基于板的测序方法相比,成本有所降低,但目前使用单细胞方法对TCR库进行分析的成本仍然高于批量方法,在某些情况下可能会受到限制。因此,未来在这些挑战上的改进对于生成更忠实的T细胞群体至关重要。例如,适应微孔板阵列技术可能提供解决TCR测序方法中这些挑战的途径,因为它们兼容更低的细胞输入,并且比基于液滴的方法更有效。
除了这些技术改进外,还有机会将这里讨论的技术与其他方法结合,以更深入地了解T细胞克隆性和特异性如何协调影响生理功能。一个具有创新潜力的领域是在体内解析T细胞的特异性和表型。虽然现有的单细胞方法可以将TCR库与表型联系起来,但T细胞在组织中的空间组织会丢失。最近开发的空间转录组技术能够在保持二维位置信息的同时进行高通量基因表达分析。对这些方法的改进可能实现空间解析的TCR和表型配对。研究TCR的克隆性和多样性在其空间背景下的能力,将对解剖T细胞及其特异性抗原的解剖分布如何塑造免疫反应起到重要作用。最后,最近在利用DNA条形码标记细胞及其后代的单细胞基因谱系追踪方法方面的进展,已被证明在阐明造血和众多其他生物过程中具有极大的价值。将谱系追踪与TCR分析相结合,有可能进一步精细化对T细胞发育轨迹和表型可塑性的理解,特别是在克隆内表型和功能异质性方面。此前追踪T细胞命运的努力依赖于携带转基因TCR的小鼠,这揭示了克隆T细胞反应的重要特征,例如一个T细胞能够产生表现出异质性分化和生长动力学模式的后代。然而,这些研究未能分析TCR克隆性和特异性在塑造T细胞反应中的作用。将TCR测序和转录组分析与基因追踪策略相结合,可以为以前所没有的分辨率在体内追踪T细胞克隆打开大门,从而实现克隆内谱系和表型层次结构的重建。尽管这些方法可能需要多次迭代才能应用于人体组织等生理环境,但此类创新仍将有助于更好地理解T细胞反应的协调方式,进而可用于开发更有效的癌症、感染和其他免疫介导疾病的治疗方法。


ImmunoDiagnostics | ImmunoMonitoring
免疫诊断 | 免疫监控
专注于免疫组高通量测序
ImmuHub | Seq-MRD | Immun-Traq
| Immun-Cheq | T-Classifier |TCR-T

